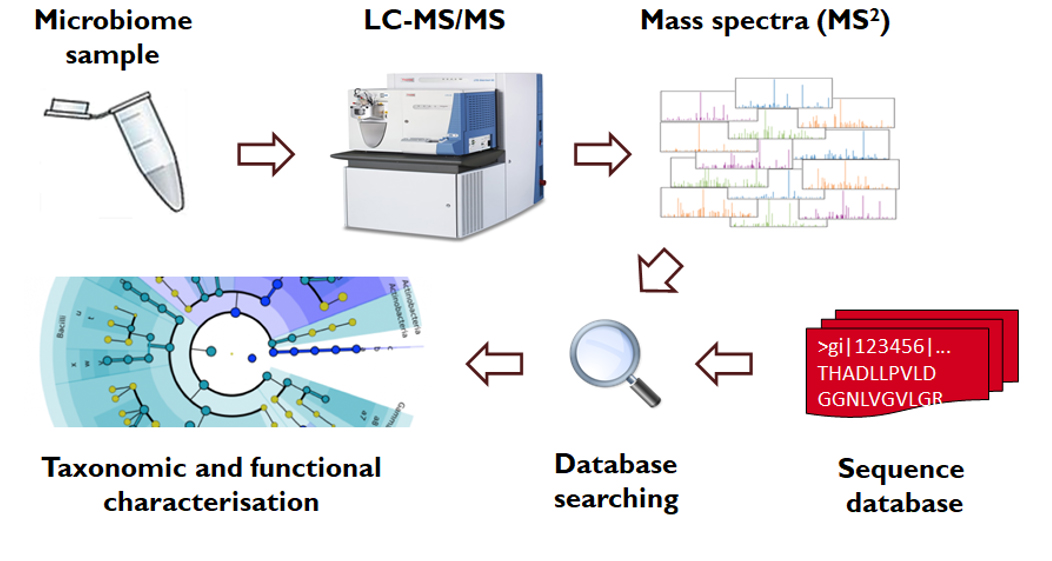
Metaproteomik-Workflow von der Probenahme über MS/MS-Erfassung bis zur Datenbank-basierten taxonomischen und funktionellen Charakterisierung
Quelle: BAM
Die Metaproteomik kann als Profilerstellung der gesamten Proteinmenge verstanden werden, die von komplexen mikrobiellen Ökosystemen wie dem menschlichen Darmmikrobiom produziert wird. Sie ermöglicht, die Funktionsweise der uns umgebenden komplexen mikrobiellen Gemeinschaften aufzudecken: Der menschliche Darm beherbergt beispielsweise viele nützliche (aber manchmal auch schädliche) Bakterien, jedoch sind mikrobielle Gemeinschaften auch im Zusammenhang mit erneuerbaren Energiequellen in Biogasanlagen wichtig. Die Metaproteomics Initiative ist ein internationales Projekt mit Mitgliedern aus Deutschland, Belgien, Norwegen, Italien und den Vereinigten Staaten. Weitere Informationen über die Initiative mit einem Kommentar in der Zeitschrift Microbiome finden Sie hier.
Die Initiative hat die Benchmarking-Studie Critical Assessment of MetaProteome Investigation (CAMPI) durchgeführt. Im Rahmen dieser Studie wurden den beteiligten Gruppen zwei verschiedene Proben zur Verfügung gestellt: ein künstliches menschliches Darmmodell und eine echte menschliche Stuhlprobe. Um Proteine und Taxa aus diesen Proben zu identifizieren und zu quantifizieren, wendeten die Labore spezifische bioanalytische Arbeitsabläufe an, mit den Schritten Probenvorbereitung, Massenspektrometrie und Bioinformatik zur Datenverarbeitung und -analyse. Anhand der jeweiligen Labormethoden erzeugten Ergebnisse bewerteten die beteiligten Labore die Auswirkungen der Arbeitsabläufe auf Anzahl und Qualität der gefundenen Peptide, Proteine, Taxa und Funktionen.
Das wichtigste Ergebnis der CAMPI-Studie ist, dass die untersuchten Arbeitsabläufe zwischen den Laboren sehr robust waren. Im Einzelnen waren die Anzahl und die Identitäten der Proteine in jedem Probentyp bei verschiedenen Protokollen und Analysen sehr ähnlich. Es wurde jedoch festgestellt, dass die Anzahl der von den einzelnen Arbeitsabläufen gefundenen Peptide recht unterschiedlich war. Diese Varianz hängt mit der unterschiedlichen Probenvorbereitung vor der Massenspektrometrie-Analyse zusammen. Obwohl die Ergebnisse in Bezug auf den Proteingehalt der Proben für jeden Arbeitsablauf ähnlich waren, zeigte die vergleichende Studie, dass kritische Schritte der Probenvorbereitung den Nachweis bestimmter Peptide und Proteine beeinflussen. Die Ergebnisse dienen als Grundlage für künftige Multi-Lab-Studien, die erforderlich sind, um einheitliche Ansätze and Standards für die Analytik in der Metaproteomik zu erarbeiten.
Critical assessment of MetaProteome Investigation (CAMPI): A multi-laboratory comparison of established workflows
T. Van Den Bossche, B. Kunath, K. Schallert ... T. Muth
veröffentlicht in Nature Communications, Band 12, Aufsatznr. 7305, 2021
BAM Fachbereich eScience


